1 Introdução
Os físicos chamam a função que representa um elétron confinado em um potencial central de função de onda. Os químicos preferem chamar essa mesma função de orbital.
É importante conhecer as duas linguagens: se expressar através das funções de onda e por meio dos orbitais. Por exemplo, a soma das funções de onda 1√2(ψ21−1−ψ211), sendo:
ψ21−1=1√64π(Za)52re−Zr/2asinθe−iϕ,
ψ211=−1√64π(Za)52re−Zr/2asinθeiϕ,
é o orbital 2px:
2px=1√32π(Za)52re−Zr/2asinθcosϕ.
Já o orbital 2py é a superposição das funções de onda i√2(ψ21−1+ψ211):
2py=1√32π(Za)52re−Zr/2asinθsinϕ.
E, o orbital 2pz é a própria função de onda ψ210, ele não é fruto de superposição:
2pz=1√32π(Za)52re−Zr/2acosθ.
Ademais, as funções de onda ψ211 e ψ21−1 são chamadas de orbitais 2p+1 e 2p−1, respectivamente (Peter Atkins 2011).
No geral, a função de onda é escrita como ψnlm, onde: n é o número quântico principal, l é o momento angular orbital, e m é a projeção do momento angular orbital no eixo z. Assim, as funções de onda ψ211, ψ210 e ψ21−1 possuem n=2, l=1 e projeções m=1,0,−1, respectivamente.
Do ponto de vista dos orbitais, os números l recebem nomes especiais, dados pela convenção de espectroscopia atômica:
l=0⟹s,l=1⟹p,l=2⟹d,l=3⟹f,l=4⟹g,l=5⟹h.
Por isso, os orbitais s são orbitais de momento angular orbital l=0, os orbitais p são orbitais de momento angular orbital l=1, e assim por diante.
Percebe-se que as equações (1.3), (1.4) e (1.5) são funções de três variáveis, pois a distância do elétron ao núcleo depende das coordenadas do elétron em relação ao núcleo:
r=√x2+y2+z2.
Os ângulos θ e ϕ também dependem das coordenadas do elétron em relação ao núcleo:
θ=arccos(z/r),ϕ=arctan(y/x).
O artigo foi escrito com o objetivo de compartilhar a técnica computacional envolvida na visualização de funções com 3 variáveis. Como todos sabem, para se graficar uma função com 3 variáveis necessitaríamos de um espaço com 4 eixos: 3 eixos para as variáveis (x,y,z), e um quarto eixo para se colocar os valores da função f(x,y,z). Fica claro que não há possibilidade de graficar esse tipo de função da mesma maneira que graficamos uma função com 2 variáveis: utilizando 2 eixos para as variáveis (x,y), e um terceiro eixo com o valor da função f(x,y).
No caso das funções com 3 variáveis, deve-se utilizar métodos de visualização volumétrica, os quais criam uma superfície no espaço, de pontos (x,y,z), sendo que, ao percorrer essa superfície, a função f(x,y,z) é uma constante. Em outras palvras, contrói-se uma superfície tridimensional na qual f(x,y,z)=α, sendo α um parâmetro. As superfícies de contorno tridimensionais assim construídas são chamadas isosuperfícies.
Vamos utilizar como método de visualização de isosuperfícies,
o algoritmo dos cubos marchando (marching cubes algorithm),
desenvolvido por Lorensen e Cline (W E Lorensen 1987).
Este algoritmo faz parte do utilitário contour3d, do pacote misc3d,
desenvolvido por Feng and Tierney para a linguagem de programação R (Dai Feng 2008).
2 Os elementos de trabalho
Antes de começarmos a fazer os gráficos dos orbitais, vamos entender os elementos de trabalho.
Estudaremos a estrutura eletrônica da molécula de hidrogênio, H2, formada por 2 núcleos de hidrogênio e por 2 elétrons. Os núcleos serão identificados por núcleo a e núcleo b, e os elétrons por elétron 1 e elétron 2.
A Fig. 2.1 mostra a geometria do problema. Os núcleos foram colocados sobre o eixo z e estão separados pela distância R. As distâncias dos elétrons i=1,2 até os núcleos a e b são representadas pelos vetores rai e rbi, de módulos:
rbi=√x2i+y2i+(zi−R)2,rai=√x2i+y2i+z2i.
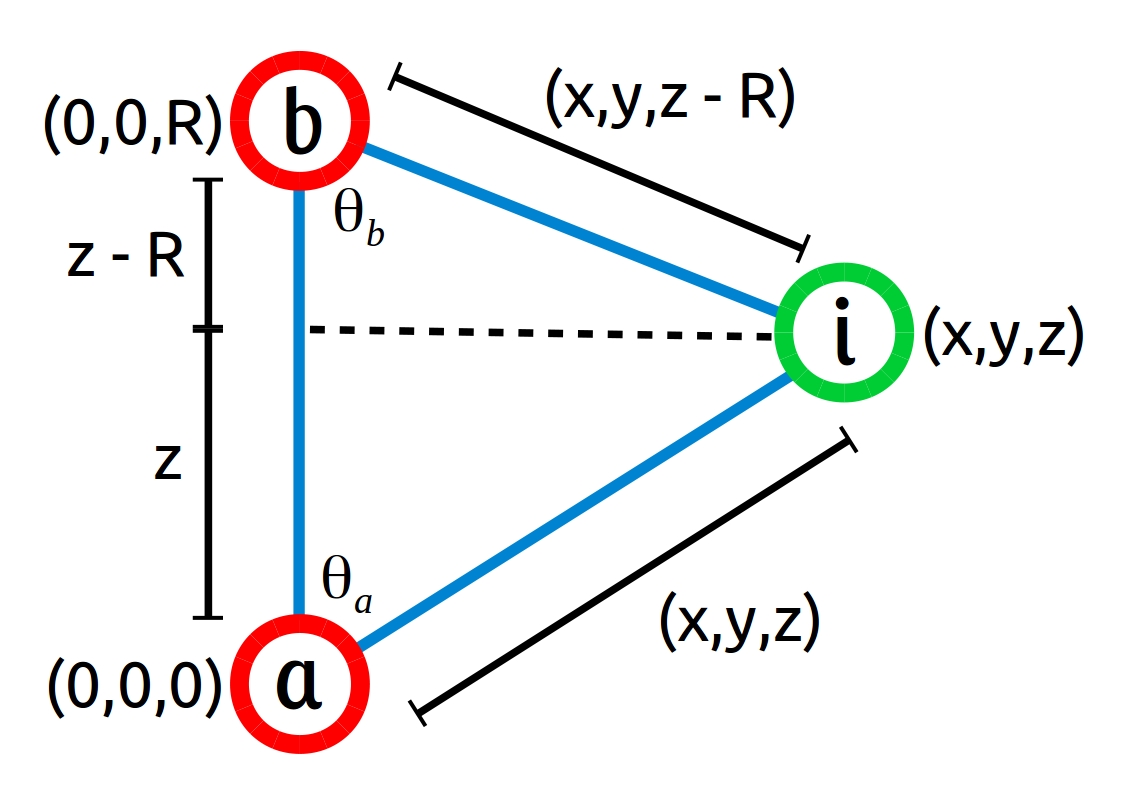
Figura 2.1: Distâncias e ângulos dos elétrons i=1,2 em relação aos núcleos a e b.
Os ângulos que os elétrons i=1,2 fazem com o eixo z, são:
θbi=arccos[(zi−R)/rbi],θai=arccos[zi/rai].
Os ângulos que os elétrons i=1,2 fazem com o eixo x, são:
ϕbi=arctan(yi/xi),ϕai=arctan(yi/xi).
3 A ocupação de orbitais
O elétron 1 pode ocupar o orbital atômico do núcleo a. Vamos representar essa ocupação dizendo que ψa(1). Se o elétron 1 está no orbital do núcleo a, é possível que o elétron 2 esteja no orbital do núcleo b, então, é possível escrever ψb(2). Esta maneira de ocupar os orbitais dos átomos de hidrogênio proporciona que o orbital da molécula de H2 seja:
Ψ=ψa(1)ψb(2).
Todavia, também é possível que o elétron 2 ocupe o orbital atômico do núcleo a e o elétron 1 ocupe o orbital do núcleo b, então, também é possível escrever ψa(2) e ψb(1), respectivamente. Esta nova configuração viabiliza que o H2 tenha o seguinte orbital molecular:
Ψ=ψa(2)ψb(1).
Visto que (3.1) e (3.2) são igualmente prováveis, o orbital molecular total do H2 deve ser a superposição das funções de onda de cada possibilidade (Peter Atkins 2011):
Ψ=ψa(1)ψb(2)+ψa(2)ψb(1).
Nota: A equação acima não está normalizada.
Vamos entender como utilizar a equação (3.3). A ideia é atribuir orbitais eletrônicos ao redor dos núcleos a e b. Vamos supor que ψa=2pz e que ψb=2pz. Substituindo a fórmula (1.5) em (3.3), e deixando de lado a constante 1√32π(1a)52, tem-se:
Ψ=ra1e−ra1/2acosθa1×rb2e−rb2/2acosθb2+ra2e−ra2/2acosθa2×rb1e−rb1/2acosθb1.
Nota: Como estamos trabalhando com hidrogênios, Z=1.
As distâncias são determinadas pelas fórmulas (2.1):
ra1=√x21+y21+z21,rb2=√x22+y22+(z2−R)2,ra2=√x22+y22+z22,rb1=√x21+y21+(z1−R)2.
Já os ângulos são calculados pelas equações (2.2):
θa1=arccos[z1/ra1],θb2=arccos[(z2−R)/rb2],θa2=arccos[z2/ra2],θb1=arccos[(z1−R)/rb1].
4 O código fonte
Estamos com as ferramentas necessárias para dar início à escrita do código que vai gerar os gráficos das isosuperfícies.
A ideia central é fazer uma malha de pontos (x,y,z)
com três comandos seq(-10,10,length=101), para alimentar as fórmulas dos orbitais.
Depois, escrever uma função com o comando function() que vai conter as fórmulas dos orbitais
e, em seguida, chamar o comando contour3d que se encarregará de fazer as isosuperfícies.
#Construção da malha espacial de pontos
x <- seq(-17,18,length=101)
y <- seq(-16,19,length=101)
z <- seq(-15,20,length=101)
#Função que contém as fórmulas dos orbitais
OrbMO <- function(x,y,z) {
x1 <- x
y1 <- y
z1 <- z
R <- 1.4
r_b1 <- sqrt(x1^2 + y1^2 + (z1-R)^2)
theta_b1 <- acos( (z1-R)/r_b1 )
phi_b1 <- atan2(y1,x1)
x2 <- x
y2 <- y
z2 <- z
r_b2 <- sqrt(x2^2 + y2^2 + (z2-R)^2)
theta_b2 <- acos( (z2-R)/r_b2 )
phi_b2 <- atan2(y2,x2)
r_a1 <- sqrt(x1^2 + y1^2 + z1^2)
theta_a1 <- acos(z1/r_a1)
phi_a1 <- atan2(y1, x1)
r_a2 <- sqrt(x2^2 + y2^2 + z2^2)
theta_a2 <- acos(z2/r_a2)
phi_a2 <- atan2(y2, x2)
b1 <- exp(-r_b1 ) /sqrt( pi) #1s
b2 <- exp(-r_b2 ) /sqrt( pi) #1s
a1 <- exp(-r_a1 ) /sqrt( pi) #1s
a2 <- exp(-r_a2 ) /sqrt( pi) #1s
b1 <- (2 - r_b1) * exp(-r_b1/2) /sqrt(32*pi) #2s
b2 <- (2 - r_b2) * exp(-r_b2/2) /sqrt(32*pi) #2s
a1 <- (2 - r_a1) * exp(-r_a1/2) /sqrt(32*pi) #2s
a2 <- (2 - r_a2) * exp(-r_a2/2) /sqrt(32*pi) #2s
b1 <- cos(theta_b1) * r_b1 * exp(-r_b1/2) /sqrt(32*pi) #2pz
b2 <- cos(theta_b2) * r_b2 * exp(-r_b2/2) /sqrt(32*pi) #2pz
a1 <- cos(theta_a1) * r_a1 * exp(-r_a1/2) /sqrt(32*pi) #2pz
a2 <- cos(theta_a2) * r_a2 * exp(-r_a2/2) /sqrt(32*pi) #2pz
b1 <- sin(theta_b1)*cos(phi_b1) * r_b1 * exp(-r_b1/2) /sqrt(32*pi) #2px
b2 <- sin(theta_b2)*cos(phi_b2) * r_b2 * exp(-r_b2/2) /sqrt(32*pi) #2px
a1 <- sin(theta_a1)*cos(phi_a1) * r_a1 * exp(-r_a1/2) /sqrt(32*pi) #2px
a2 <- sin(theta_a2)*cos(phi_a2) * r_a2 * exp(-r_a2/2) /sqrt(32*pi) #2px
a1 #Fórmula do orbital atômico
a1 * b2 + a2 * b1 #Fórmula do orbital molecular
}
contour3d(OrbMO, x,y,z, level=0.001) #Construção da isosuperfícieO programa é bem flexível,
pode-se comentar linhas para gerar isosuperfícies das mais variadas:
observe que há o bloco do orbital 1s, 2s, 2pz e 2px.
Lembrando que em R, os comentários são feitos com #.
Há duas opções de gráficos: (1) pode-se graficar o orbital atômico, deixando a linha a1 ativa;
ou (2) pode-se graficar o orbital molecular, deixando a linha a1*b2+a2*b1 ativa.
É o vamos fazer a partir de agora.
5 Os orbitais atômicos
Para graficar os orbitais atômicos, deve-se comentar a linha a1*b2+a2*b1.
Ademais, deve-se deixar ativo apenas o bloco do orbital que se deseja graficar.
Por exemplo, para graficar o orbital atômico 1s,
deve-se comentar os blocos 2s, 2pz, 2px
(colocando # na frente de cada linha).
As figuras 5.1, 5.2, 5.3 e 5.4, apresentam os orbitais atômicos 1s, 2s, 2pz e 2px do hidrogênio, respectivamente.
Por causa da fórmula do orbital 2s apresentar o termo (2−r), seu gráfico mostra duas cascas, enquanto que o gráfico do orbital 1s apresenta somente uma.
Na visualização dos orbitais 2pz e 2px,
foi utilizada a técnica de disparar várias isosuperfícies concentrícas.
Isso é feito atribuindo valores à opção level do comando contour3d.
Como o máximo e o mínimo desses orbitais ocorrem entre (−0,07 e 0,07),
foi escolhido os seguintes valores:
cut <- c(-0.06, -0.04, -0.02, 0.02, 0.04, 0.06)
contour3d(OrbMO, x,y,z, level=cut)Nota: Confira no artigo (Dai Feng 2008)
mais exemplos de como utilizar o pacote misc3d e seu poderoso comando contour3d.
Fica claro que o orbital 2pz aponta para o eixo z, e o 2px para o eixo x.
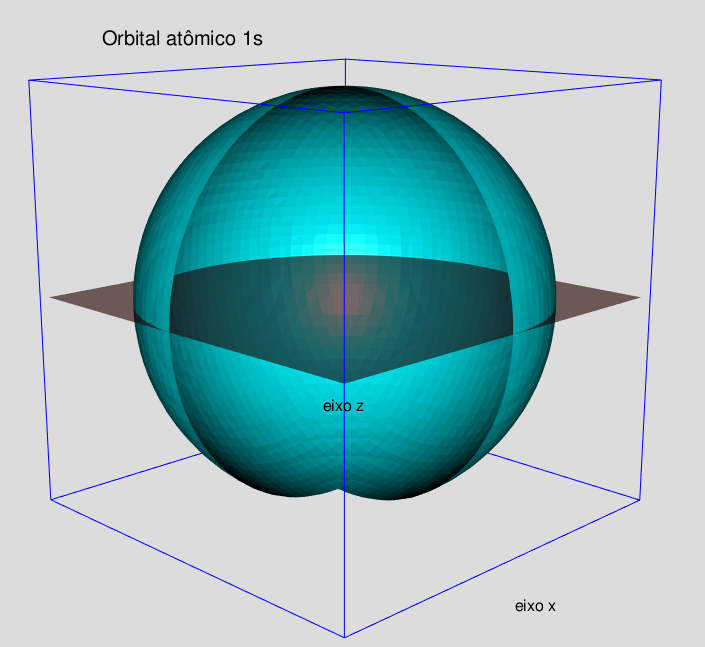
Figura 5.1: Núcleo de hidrogênio exibindo orbital 1s.
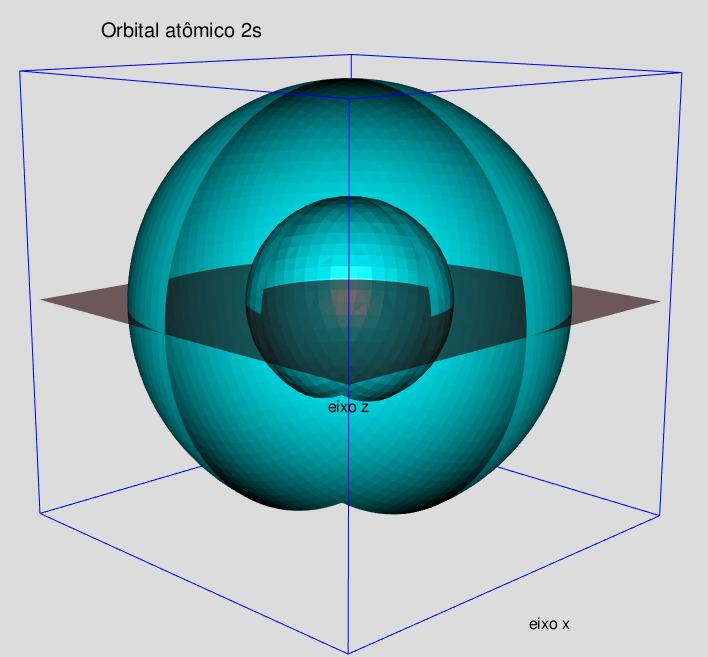
Figura 5.2: Núcleo de hidrogênio exibindo orbital 2s.
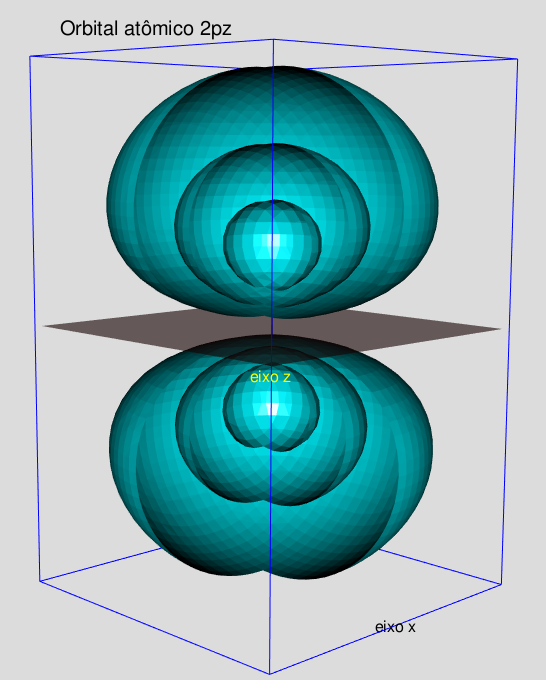
Figura 5.3: Núcleo de hidrogênio exibindo orbital 2pz: 3 isosuperfícies.
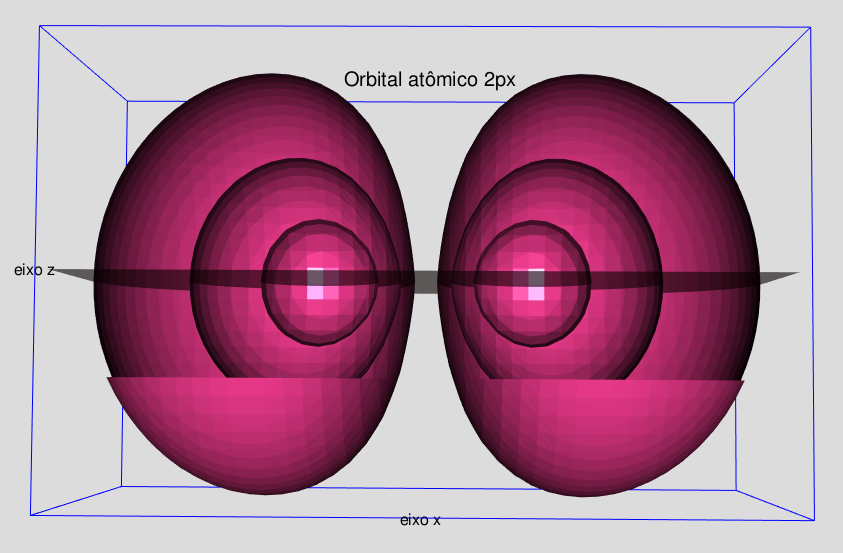
Figura 5.4: Núcleo de hidrogênio exibindo orbital 2px: 3 isosuperfícies.
6 Os orbitais moleculares
Agora, para graficar os orbitais moleculares, deve-se comentar a linha a1.
Ademais, deve-se novamente deixar ativo apenas o bloco do orbital que se deseja graficar.
O valor experimental da distância entre os núcleos a e b, da molécula de hidrogênio, é R=1,40 a0, segundo (Peter Atkins 2011).
Nota: Os orbitais estão sendo graficados ajustando a0=1 para efeito de simplificação.
As figuras 6.1, 6.2, 6.3 e 6.4, apresentam os orbitais moleculares da molécula de hidrogênio, com os núcleos a e b exibindo: ambos 1s (1s1s); ambos 2s (2s2s); ambos 2pz (2pz2pz); e ambos 2px (2px2px).
Nota: Os casos 2pz2pz e 2px2px apresentam 3 isosuperfícies concentrícas.
Planos cortando a localização dos núcleos também foram graficados: para o núcleo a, o plano é (x,y,0), e para o núcleo b, (x,y,R). Os comandos para graficar os planos são:
planes3d(0,0,1, d=-1.4, col="red", alpha=0.6) #Plano sobre o núcleo "b"
planes3d(0,0,1, d= 0.0, col="red", alpha=0.6) #Plano sobre o núcleo "a"O orbital molecular do caso 1s1s apresenta apenas 1 casca, enquanto que o do caso 2s2s, 3 cascas: decorrente da multiplicação de termos (2−ra) e (2−rb).
A ligação efetuada pelo orbital molecular 2pz2pz é predominantemente na direção do eixo z, enquando que a do 2px2px é na direção do eixo x.
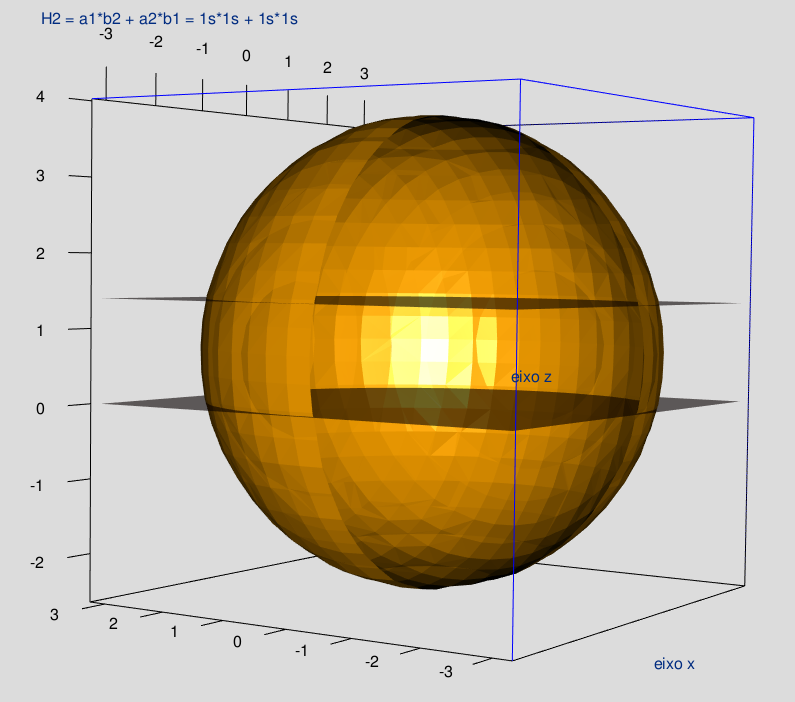
Figura 6.1: H2: Núcleos a e b na configuração 1s1s.

Figura 6.2: H2: Núcleos a e b na configuração 2s2s.
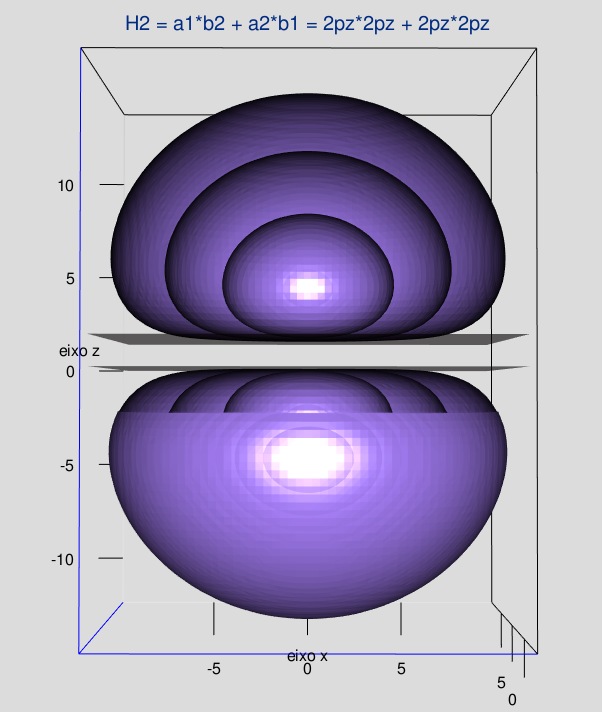
Figura 6.3: H2: Núcleos a e b na configuração 2pz2pz: 3 isosuperfícies.
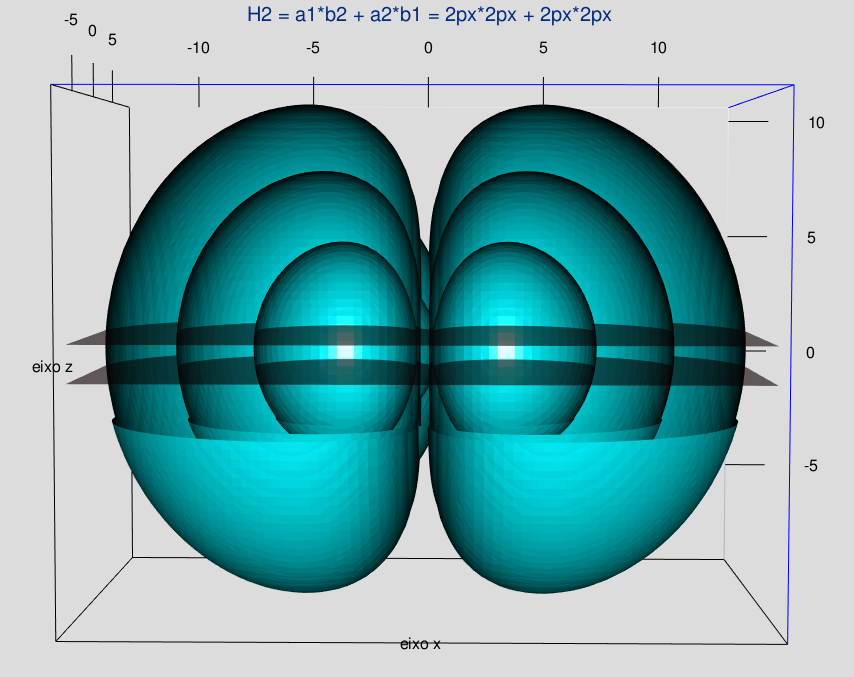
Figura 6.4: H2: Núcleos a e b na configuração 2px2px: 3 isosuperfícies.
Vale ressaltar que, em um ambiente natural, é atípico a molécula de hidrogênio estar excitada a ponto de seus elétrons ocuparem o orbital molecular 2pz2pz, ou 2px2px. É comum que ela se encontre no estado fundamental: quando os 2 elétrons ocupam o orbital molecular 1s1s. Já em um ambiente agitado, a molécula de hidrogênio pode ir para o primeiro estado excitado: construído com um elétron ocupando o orbital atômico 1s, e o outro elétron, o 2s, resultando na configuração molecular 1s2s. Este orbital misto também é possível de ser graficado acrescentando o seguinte bloco ao código fonte (ver figura 6.5):
b1 <- (2 - r_b1) * exp(-r_b1/2) /sqrt(32*pi) #2s
b2 <- (2 - r_b2) * exp(-r_b2/2) /sqrt(32*pi) #2s
a1 <- exp(-r_a1 ) /sqrt( pi) #1s
a2 <- exp(-r_a2 ) /sqrt( pi) #1s
Figura 6.5: Molécula de hidrogênio na configuração 1s2s.
7 Conclusão
A visualisação de funções com 3 variáveis é um problema gráfico que exige técnicas computacionais avançadas.
Com a pesquisa do artigo (Dai Feng 2008),
pude notar que seria possível visualizar orbitais atômicos e moleculares utilizando a linguagem R.
O código fonte compartilhado é flexível e pode ser implemantado para outras situações.
